Auricher Wissenschaftstage –
Forum einer dritten Kultur
Praktikum am Max-Planck-Institut für molekulare Genetik in Berlin
vom 19. bis zum 30. Oktober 2015
Von Insa Bohlen und
Imke Neelen
Im Rahmen der Auricher Wissenschaftstage erhielten wir die Möglichkeit, während eines zweiwöchigen Praktikums vom 19.10. bis zum 30.10.2015 am Max-Planck-Institut für molekulare Genetik in Berlin einen Einblick in die Forschung zu gewinnen.
Das Max-Planck-Institut für molekulare Genetik ist eines der führenden Genomforschungszentren weltweit und gehört zu den größten Einrichtungen innerhalb der Max-Planck-Gesellschaft. Unter anderem werden die genetischen Ursachen geistiger Behinderungen, das funktionelle Zusammenspiel molekularer Netzwerke sowie Mechanismen der genetischen Regulation analysiert.
Die Forschungsgruppe „Mundlos“, in deren Arbeit wir die zwei Wochen lang Einblicke gewinnen durften, beschäftigt sich dagegen in erster Linie mit der Entstehung angeborener Skelettfehlbildungen.
Während unseres Praktikums lernten wir grundlegende Methoden der Genetik kennen. In der ersten Woche bestand unsere Hauptaufgabe darin, transgene Mäuse zu genotypisieren, indem wir zunächst die genomische DNA (gDNA) der Mäuse aus einer Ohrbiopsie extrahierten. Anschließend führten wir mit dieser DNA eine PCR (Polymerase Kettenreaktion) durch, um diese zu amplifizieren (vervielfältigen). Dazu pipettiert man zunächst zu den genomischen DNAs (Proben/Template) Wasser, Puffer mit Magnesium-Ionen (Mg2+), zwei spezifische Primer, dNTP’s und eine DNA-Polymerase (Taq-Polymerase). Die eigentliche PCR-Reaktion findet dann im Thermocycler statt, wo man die Dauer und Temperatur der folgenden Zyklen einstellen kann.
Generell wird eine PCR in drei verschiedene Abschnitte eingeteilt. (1) Denaturierung der DNA, (2) Annealing der Primer und (3) Elongation des spezifischen PCR-Produktes.
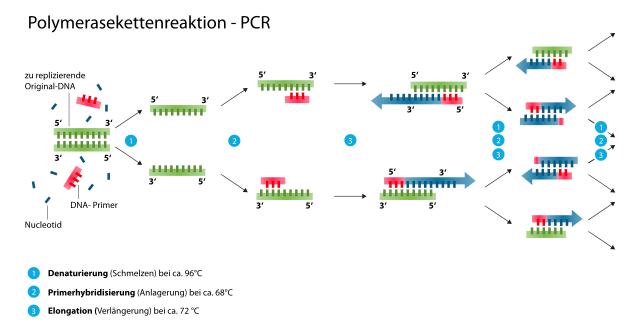
Die Phasen der PCR-Reaktion (Quelle: „Polymerasekettenreaktion“ von Enzoklop. Lizenziert unter CC BY-SA 3.0 über Wikimedia Commons.)
Als Erstes wird die DNA bei 96°C denaturiert bzw. aufgeschmolzen (1). Daraufhin kommt es bei etwa 54-60°C zur Primer Bindung am 3‘ Ende der Gensequenz (2). Bei 72°C vervielfältigt die Polymerase die gebundene DNA Sequenz (3).
Dieser Vorgang wird mehrmals wiederholt, sodass die Menge der DNA-Fragmente exponentiell zunimmt. Anschließend werden die PCR-Produkte in einer Agarose-Gelelektrophorese ihrer Größe entsprechend aufgetrennt. Dazu werden die PCR-Produkte in die Taschen des vorbereiteten Gels geladen, welches in einer Laufkammer an Spannung angeschlossen wird. Dadurch bewegt sich die negativ geladene DNA in Richtung Anode (+). Dabei bewegen sich kürzere Moleküle weiter durch das Gel als längere, wodurch die unterschiedlichen Banden in der unteren Abbildung zustande kommen.
Die Visualisierung der DNA erfolgt durch den Zusatz eines DNA-interkalierenden Farbstoffes (Ethidiumbromid).
In der Abbildung links ist eine typische Genotypisierungs-PCR dargestellt. Die oberen (größeren) Banden sind die Mutantenbanden und die unteren (kleineren) die Wildtypbanden, sodass eine Maus mit einer Bande unten ein Wildtyp ist, eine Maus mit einer Bande oben ein homozygoter Mutant und eine Maus mit beiden Banden ein heterozygoter Mutant. Die letzten drei Spalten stellen Kontrollproben dar, um sicher gehen zu können, dass die erzielten Ergebnisse richtig sind. Bei der NTC (No Templet Control) handelt es sich um Wasser. Somit sollte an dieser Stelle keine Bande zu sehen sein, weil die Ergebnisse sonst durch eine Kontamination verfälscht sein könnten.
Des Weiteren haben wir gelernt, Embryonenschnitte anzufertigen, indem wir den vorfixierten Embryo in eine kleine Form mit einem Wachs-Alkoholgemisch gaben und anschließend bei -80°C gefroren. So kann man nun im Cryotom 8μm dicke sagittale Schnitte vom Embryo erstellen und unter dem Mikroskop betrachten. Auf diese Weise kann man die Entwicklung des Embryos verfolgen oder auf bestimmte Enzyme oder Genexpressionen untersuchen.
Darüber hinaus lernten wir, wie man die DNA genetisch manipuliert, indem man in ein Plasmid das gewünschte Gen integriert und kloniert.
Zunächst extrahiert man dazu die genomische DNA, indem man als Erstes die Zellwände zerstört und die DNA reinigt. Anschließend fällt man die DNA mit Hilfe von Salzen und Alkoholen und wickelt sie auf. Nun lässt man sie über Nacht trocknen und resuspendiert sie am folgenden Tag wieder in Wasser.
Auf dieser extrahierten genomischen DNA macht man nun eine PCR, um das spezifische DNA-Fragment zu erhalten. Da die Größe des Gens bekannt ist, kann man es nach einer Gelelektrophorese identifizieren, ausschneiden und aufreinigen.
In der anschließenden Ligation verbinden Ligasen die DNA-Fragmente unter ATP-Verbrauch mit Plasmiden, welche bereits eine bestimmte Antibiotikaresistenz besitzen. Diese Plasmide werden anschließend in Bakterienzellen transformiert, welche auf Agarplatten mit dem entsprechenden Antibiotikum ausgestrichen werden. So können nur die Bakterienzellen überleben, die das Plasmid aufgenommen haben.
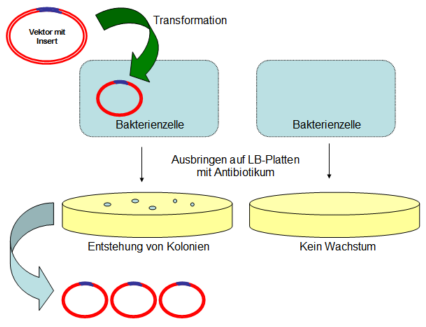
Transformation eines Plasmids in eine Bakterienzelle und anschließende Antibiotika-Selektion (Quelle: Wikipedia)
Doch um sicher zu gehen, dass das Plasmid auch das DNA-Fragment eingebaut hat, pickt man gewachsene Kolonien von der Agarplatte und führt eine weitere Kolonie-PCR durch. Positive Kolonien werden dann über Nacht zu Bakterienkulturen angereichert und anschließend präpariert. Das heißt, die Zellen der Bakterien werden lysiert und an eine Säule gebunden.
Daraufhin wird die DNA auf der Säule gewaschen und anschließend eluiert (herausgelöst), sodass nun pure Plasmid-DNA vorliegt. Diese kann man nun z. B. in Stammzellen transfizieren. Über verschiedene molekulare Mechanismen kann die transfeszierte DNA in das Genom der Stammzellen aufgenommen werden. Die somit veränderten Stammzellen können dann untersucht werden, indem man sie z. B. zu Nervenzellen differenziert oder aus ihnen Mäuse generiert. Die zuvor durchgeführte Klonierung der DNA kann so z. B. zu unterschiedlicher Genexpression führen, die Aufschluss über Genregulation geben kann.
Abschließend können wir sagen, dass das Praktikum am Max-Planck-Institut sehr interessant war. In diesen zwei Wochen haben wir sehr viel über die Arbeit in der Forschung gelernt und wichtige Erfahrungen dazu gewonnen. Es machte uns außerdem sehr viel Spaß, da wir viele Aufgaben selbst übernehmen durften, und wir waren positiv überrascht, wie viel Zeit sich die Forscher für uns nahmen, um uns Fragen zu beantworten und Vorgänge zu erklären.
So bedanken wir uns bei der Forschungsgruppe „Mundlos“, insbesondere bei Jürgen Stumm, Manuel Cosme, Asita Carola Stiege und Alexandra Despang für die Betreuung und besonders bei den Organisatoren der Auricher Wissenschaftstage, die uns dieses Praktikum ermöglicht haben.