Auricher Wissenschaftstage –
Forum einer dritten Kultur
Praktikum am Max-Planck-Institut für molekulare Genetik in Berlin
vom 25. März bis zum 6. April 2007
Von Lene Fabricius
Im Rahmen der Auricher Wissenschaftstage habe ich die Möglichkeit erhalten, ein zweiwöchiges Praktikum am Max-Planck-Institut für molekulare Genetik in Berlin zu absolvieren. In dieser Zeit wurde ich in der Abteilung von Prof. Hans Lehrach aufgenommen, in welcher etwa 100 Mitarbeiter des Instituts beschäftigt sind. Ich arbeitete an dem von der EU geförderten Projekt EURExpress innerhalb der Arbeitsgruppe von M.-L. Yaspo mit. Weiter habe ich auch kurze Einblicke in andere Projekte gewinnen können, die sehr interessant und beeindruckend waren.

Das Max-Planck-Institut für molekulare Genetik in Berlin
EURExpress ist ein Hochdurchsatzprojekt, welches 12 wissenschaftliche Institutionen aus Italien, Frankreich, Spanien, England und Deutschland mit einbezieht. Es ist auf insgesamt vier Jahre ausgelegt und strebt an, Genexpressionsdaten für alle Mausgene (~ 20.000) zu erfassen. Es soll letztendlich ein sogenannter Transkriptomatlas erstellt werden, der die Aktivität einzelner Gene in Mausembryonen beschreibt. Dieses wird mittels eines semi-automatisierten experimentellen Verfahren erzeugt, das auch RNA in situ-Hybridisierung genannt wird.
Während meines Praktikums durfte ich die einzelnen Schritte des Projektes mit verfolgen und teilweise selbst ausführen.
Nachdem die Embryonen aus der Maus heraus präpariert und in ein spezielles Medium gebettet worden sind, werden sie mittels eines Mikrotoms – ein Gerät, das anhand einer sehr scharfen Klinge es erlaubt, hauchdünne Schnitte zu erzeugen (bis zu 4 µm (1.000 µm = 1mm)) – in 25 µm dünne sagitale Scheiben geschnitten und auf Objektträger ausgerichtet. Diesen Vorgang konnte ich selbst ausführen.
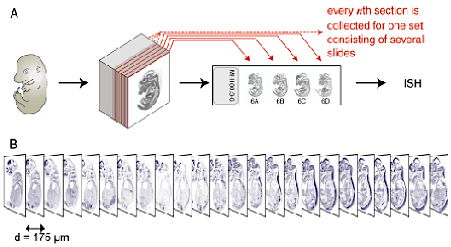
Abbildung aus Visel et al., Nucleic Acid Research, 2004

Beim Schneiden am Mikrotom
Anschließend werden die Schnitte „fixiert“, ein Verfahren, mit dem die Schnitte fest an den Objektträger gebunden werden. Dieses ermöglicht eine lange Aufbewahrungszeit. Zusammenfassend werden die Schnitte nach einem festgelegten Protokoll in einem „Stainer“ automatisch in verschiedene Lösungen getaucht und sind nach mehreren Durchläufen auf den Objektträgern fixiert. Sie können sich nun nicht mehr ablösen.
Nach dem Fixieren unterliegen die Schnitte einer Qualitätskontrolle mit einem Mikroskop. Unsaubere Schnitte, die nicht verwertbar sind, werden aussortiert. Bei der Qualitätskontrolle habe ich selbst mitgewirkt und die Ergebnisse anschließend in der lokalen Datenbank erfasst.

Ein Objektträger in einer Kammer nach dem ISH Vorgang (dunkelblaue Färbung) [Quelle: EURExpress]
Nun folgt der wichtigste Schritt, die in situ-Hybridisierung (ISH). Bei diesem Verfahren werden einzelne Nukleinsäuren (RNA) in Zellen oder Geweben nachgewiesen. Dafür wird eine künstlich hergestellte Sonde aus markierten Nukleinsäuren eingesetzt, die über Basenpaarungen an die nachzuweisende Nukleinsäure (RNA) „hybridisiert“, also bindet. Dieses Verfahren findet in einem speziell dafür entwickelten Roboter statt, in dem gleichzeitig 384 Schnitte bearbeitet werden können. Dazu werden die Objektträger in Kammern eingehängt, welche dann im Roboter eingesetzt werden. In einem 24-Stunden-Protokoll werden anschließend unterschiedliche Lösungen durch die Kammern gebracht, einschließlich der markierten Sonden. Die Aktivität eines Gens wird während eines Färbungsschrittes sichtbar gemacht und lässt in dunkelblauer Farbe erkennen, in welchem Gewebe und/oder Zelltyp das Gen aktiv ist. Die ISH wird zwei bis dreimal Mal pro Woche für das EU-Projekt durchgeführt. In den vier Jahren, die hierfür angesetzt worden sind, soll die Aktivität von etwa 20.000 Genen erforscht werden.
Hier konnte ich die Schnitte, die ich selbst angefertigt habe, mitlaufen lassen, um die Aktivität eines sogenannten Kontrollgens zu untersuchen.
Anschließend werden die Schnitte getrocknet, damit sie erneut mikroskopiert und ausgewertet werden können. Nach einer Qualitätsbestimmung werden die „guten“ Schnitte digitalisiert. Dies wird anhand eines Mikroskops gemacht, das speziell für dieses Projekt entwickelt wurde.

Das Scanning-Mikroskop [Quelle: EURExpress]
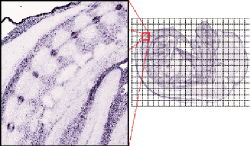
Eine digitalisierte Aufnahme des Scanning-Mikroskops [Quelle: EURExpress]
Die Bilder werden automatisch abgelichtet, gespeichert und bearbeitet. Anschließend werden die Genexpressionsdaten in einer Datenbank in England hochgeladen. Dort werden die Expressions-Muster genau beschrieben und dann auf einer Webseite publiziert <www.eurexpress.org>. Diese ist dann frei zugänglich, und Wissenschaftler aus aller Welt können sich Informationen daraus entnehmen.
Da sich die Gene der Maus mit denen eines Menschen vergleichen lassen, dient dieses Projekt auch dazu, Informationen zur Erforschung von menschlichen Krankheiten zu gewinnen.
Ferner konnte ich Einblicke in den Bereich von Zellkulturen gewinnen. Zellkulturen sind ein wichtiger Bestandteil in Forschungseinrichtungen; sie ermöglichen die Untersuchung verschiedener Prozesse in lebenden Strukturen. In der Chromosom 21-Arbeitsgruppe werden den Zellen beispielsweise bestimmte Wachstumsfaktoren hinzugefügt, wodurch deren Teilung angeregt wird. Die Zellen wachsen auf einem Nährboden und müssen nach einer bestimmten Zeit aufgeteilt und auf einen neuen Nährboden umgesetzt werden, damit ihre Dichte nicht zu groß wird. Durch diese Aufteilung der Zellen, auch „passagieren“ genannt, können Zelllinien fast beliebig lang erhalten bleiben. Auch hier konnte ich selbständig mitwirken und die Schritte eigenständig ausführen.

Beim Pipettieren in der Zellkultur
Einige Zellen habe ich auf einem Objektträger fixiert und anschließend die Lokalisierung eines bestimmten Proteins mittels Fluoreszenzmikroskopie nachgewiesen. Nach einem vorgegebenen Protokoll werden den Zellen bestimmte Lösungen, Farbstoffe und eine markierte Sonde (hier Antikörper gekoppelt mit einem Fluoreszenzmolekül) hinzugefügt. Unter dem Mikroskop lassen sich dann sowohl die Zellkerne deutlich vom Cytoplasma unterscheiden als auch die Lokalisierung des Proteins X erkennen.

Am Fluoreszenzmikroskop

Zellaufnahmen mit dem Mikroskop
Auch alltägliche Abteilungsarbeiten wie das „Autoklavieren“ (Sterilisieren von Geräten und Materialien) oder Destillieren von Wasser durfte ich mir ansehen.
Abschließend kann ich sagen, dass das Praktikum am Max-Planck-Institut sehr interessant war. Ich habe in diesen zwei Wochen vieles gelernt und einen bleibenden Eindruck erhalten. Es hat großen Spaß gemacht, und ich war erstaunt, wie viele Aufgaben ich selbst übernehmen durfte und welche verantwortungsvollen Schritte mir zugetraut wurden. Ich bedanke mich bei Herrn Stracke und Frau Dr. Falter, die mir dieses Praktikum im Rahmen der Auricher Wissenschaftstage ermöglicht haben. Weiter gilt ein besonderer Dank der Chromosom 21-Arbeitsgruppe (Dr. Ilaria Piccini, Dr. Marc Sultan und Dr. Marie-Laure Yaspo), die sich mit sehr ausführlichen und verständlichen Erklärungen ausreichend Zeit für mich genommen hat.